Membranproteinfaltung, Stabilität und Funktionen
Yu-Chu Chang
Wir untersuchen die biophysikalischen und biochemischen Eigenschaften von Membranproteinen, mit dem Schwerpunkt auf ihrer Faltung, Stabilität und Funktion. Dabei setzen wir eine Vielzahl von Techniken ein, darunter neuartige Methoden zur Untersuchung der Membranproteinstabilität sowie biochemische Assays zur Charakterisierung der Funktion von bakteriellen Virulenzproteinen, wie beispielsweise des Wzx-Proteins in der Lipopolysaccharid-Biosynthese.
Entwicklung neuartiger Techniken zur Untersuchung der Membranproteinfaltung und -stabilität
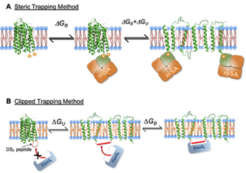
Unsere Forschung konzentriert sich auf die biophysikalischen und biochemischen Eigenschaften helikaler Membranproteine, insbesondere deren Faltungsstabilität und -kinetik. Wir entwickeln neue Methoden zur Untersuchung der Membranproteinstabilität in Lipiddoppelschichten und unter Lipid-Disc-Bedingungen.
Bacteriorhodopsin (bR) dient uns als Modellmembranprotein zur Entwicklung verschiedener neuer Methoden. Die Techniken der sterischen Inhibition und des "clipped trapping" nutzen die Kopplung der Faltung des Zielmembranproteins an ein Bindungsereignis. Die Faltung/Entfaltung des Membranproteins kann durch die Bindung eines anderen löslichen Proteins induziert werden. Diese Techniken zielen darauf ab, die Faltungseigenschaften von Membranproteinen in einer physiologisch relevanteren Lipiddoppelschichtumgebung zu untersuchen, anstatt unter denaturierungsbedingten Faltungs-/Entfaltungsbedingungen.
Untersuchung von Virulenz-assoziierten Proteinen in Salmonellen
- Wzx: Dieses integrale Membranprotein ist an der Lipopolysaccharid (LPS)-Biosynthese gramnegativer Bakterien beteiligt und fungiert als Lipid-gekoppelte Oligosaccharid-Flippase in diesem Stoffwechselweg.
- YqiC: Durch Protein-Protein-Interaktionsstudien haben wir herausgefunden, dass die Funktion von YqiC eng mit dem Energiestoffwechsel der Bakterien verbunden ist, insbesondere mit der Elektronentransportkette (ETC).
Mit kombinatorischen Ansätzen untersuchen wir die strukturellen Eigenschaften, Funktionsmechanismen und Interaktionsnetzwerke dieser Proteine. Da die Knockout-Stämme dieser Gene die Kolonisations- und Invasionsfähigkeit von Salmonellen erheblich beeinträchtigen, stellen sie potenzielle Zielstrukturen für die Entwicklung neuartiger Antibiotika dar.
