Projekte
Adaptive Evolution filamentöser Pflanzenpathogene
Genomevolution
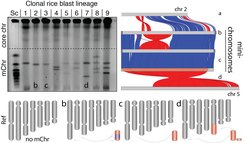
Wir haben kürzlich entdeckt, dass Magnaporthe oryzae (syn. Pyricularia oryzae), über einen sehr diversen Satz von Mini-Chromosomen, die in manchen, aber nicht allen Isolaten vorhanden sind, verfügt. Diese Mini-chromosomen tragen zum horizontalen Chromosomentransfer und umfangreichen, strukturellen Genomvariationen bei. In unserem Forschungsprogramm möchten wir folgende Fragen zur Evolution des Genoms von Pflanzenpathogenen untersuchen: Wie divers sind Mini-Chromosomen in M. oryzae Populationen?
- Wie dynamisch sind Mini-Chromosomen?
- In welchem Maße beeinflussen Mini-Chromosomen die Genomstruktur?
- Wie werden die Entstehung und der Erhalt von Mini-Chromosomen reguliert?
- Welche genomischen Elemente und molekularen Mechanismen liegen der Entstehung, Rekombination und Transfers von Mini-Chromosomen zugrunde?
- In welchem Maße beeinflusst der horizontale Transfer von Mini-Chromosomen die Genom-Evolution?
Molekulare Evolution
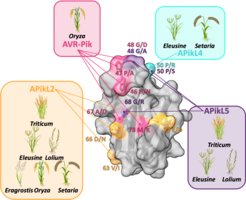
Pathogene Virulenzeffektoren evolvieren sehr schnell, um sich an verschiedene Wirtspflanzengenotypen oder -arten anzupassen. Anhand Analysen der Genomstruktur in M. oryzae Popolationen, sehen wir, dass somatische Rekombinationsevents (einschließlich mit Mini-Chromosomen assoziierter Rekombination) in genomischen Regionen auftreten, die reich an Transposons und Virulenzeffektorgenen sind. Wir möchten verstehen, in welchem Maße strukturelle, genomische Rekombinationen zu erhöhten Evolutionsraten in Virulenz-assoziierten Regionen in Pathogen-Genomen beitragen. Anschließend werden wir sowohl bioinformatisch als auch in Laborexperimenten erforschen, welche Auswirkungen diese Ereignisse auf die molekulare Evolution von Effektorproteinen haben. Letztendlich möchten wir verstehen, wie diese Mutationen die Proteinstruktur und ihre Interaktionen mit Pflanzenproteinen beeinflussen und wie diese Veränderungen den Pathogenen dabei helfen, Krankheiten auf verschiedenen Wirtspflanzen zu verursachen.
Pflanzenimmunität
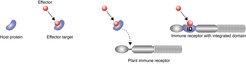
Pflanzen verfügen über ein hochkomplexes Immunsystem, dass sowohl Immunrezeptoren auf der Zelloberfläche als auch intrazelluläre Rezeptoren umfasst. In den letzten Jahren ist ein neues Konzept entstanden, das zeigt, dass bestimmte Proteindomänen, die Ziel von Pathogenangriffen sind, in pflanzlichen Immunrezeptoren integriert sind. Dort fungieren sie als sogenannte integrierte Domänen, die Pathogeneffektoren binden und so eine Immunantwort auslösen. Wir werden auf diesem Konzept aufbauen und unser Wissen aus der Erforschung der Wechselwirkungen zwischen Pathogeneffektoren und Wirtszellzielproteinen nutzen, um pflanzliche Immunrezeptoren mit neuen und verbesserten Erkennungsspezifitäten zu entwickeln.
ERC-gefördertes Projekt "PANDEMIC"
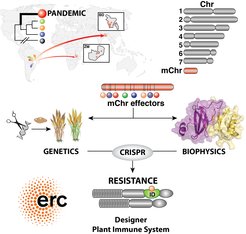
Unser Forschungsprogramm hat kürzlich eine Förderung vom Europäischen Forschungsrat (ERC) erhalten. "PANDEMIC" ist ein ehrgeiziges Projekt, um innovative, durch Wirts-Pathogen Interaktion inspirierte Strategien für die Resistenz von Nutzpflanzen gegen Pflanzenkrankheiten zu entwickeln. Die Pandemie von M. oryzae auf Weizen stellt eine klare und aktuelle Gefahr für die globale Lebensmittelversorgung dar. Sie wird durch eine bestimmte genetische Linie von M. oryzae verursacht, der erstmals vor nur etwa ~35 Jahren in Brasilien auftrat und sich in den letzten 7 Jahren nach Südostasien und Afrika ausgebreitet hat. Wir haben entdeckt, dass alle Isolate der pandemischen Linie zu einer einzigen, klonalen Gruppe gehören. Diese pandemischen Isolate tragen ein überzähliges Mini-Chromosom (mChr) von etwa 2 Mb, das 19 sekretierte Virulenzeffektoren kodiert und die pandemische Linie hochvirulent macht. Wir stellen die Hypothese auf, dass dieses mChr auch die Achillesferse der pandemischen Pilzlinie ist. In "PANDEMIC" werden wir die Virulenzeigenschaften des mChr durch Genomeditierung und biotechnologisches Design von pflanzlichen Immunrezeptoren zum Ziel machen, um die mChr-kodierten Effektoren zu erkennen und zu bekämpfen.
Das Modellsystem
Der Pilz Magnaporthe oryzae (syn. Pyricularia oryzae) verursacht die Blast-Krankheit, eine der verheerendsten Nutzpflanzenkrankheiten weltweit, die zu erheblichen Ertragseinbußen führt und eine Bedrohung für die globale Ernährungssicherheit darstellt. M. oryzae kann mehr als 50 Gras-Arten infizieren, einschließlich Reis, Gerste, Weizen, Borstenhirse und Fingerhirse. Darüber hinaus hat M. oryzae die Fähigkeit, Wirtswechsel oder Ausweitungen des Wirtsspektrums durchzuführen, was sein epidemisches Potenzial erhöht. In den 1980er Jahren trat die Krankheit erstmals auf Weizen in Brasilien auf und hat sich seitdem in großen Teilen Südamerikas und in den letzten Jahren in Südasien und Afrika verbreitet. M. oryzae verbreitet sich hauptsächlich asexuell, und klonale Linien dominieren die durch M. oryzae ausgelösten Epidemien. Unter Laborbedingungen kann jedoch sexuelle Reproduktion erreicht werden, und es wurden in natürlichen Populationen Hinweise auf Rekombination beobachtet. M. oryzae ist das ideale Modellsystem, um die Koevolution von Pflanzen und Pathogenen zu untersuchen, indem wir die Genom-Evolution, strukturelle Variation und molekulare Evolution von Virulenzeffektorgenen kombinieren. Der Pilz besitzt ein kleines (~45 Mb), haploides Genom, umfangreiche genomische Ressourcen stehen zur Verfügung, er ist genetisch zugänglich und in vitro kultivierbar und kann unter Laborbedingungen Wirtspflanzen infizieren. Sowohl der sexuelle als auch der asexuelle Zyklus können unter Laborbedingungen vollständig durchlaufen werden. Darüber hinaus ermöglichen jüngste Fortschritte in Long-Read-Sequenziertechnologien die Durchführung von Hochdurchsatz-Analysen struktureller Genomvariationen. Dies ermöglicht es uns, unsere Analysen auf eine Populationsebene auszuweiten und die Mechanismen der Genom-Evolution experimentell im Labor zu untersuchen.





